Decision Tree 결정트리
Decision Tree란?
- Tree를 만들기 위해 예/아니오 질문을 반복하며 학습한다.
- 다양한 앙상블(ensemble) 모델이 존재한다 (RandomForest, GradientBoosting, XGBoost, LightGBM)
- 분류와 회귀에 모두 사용 가능
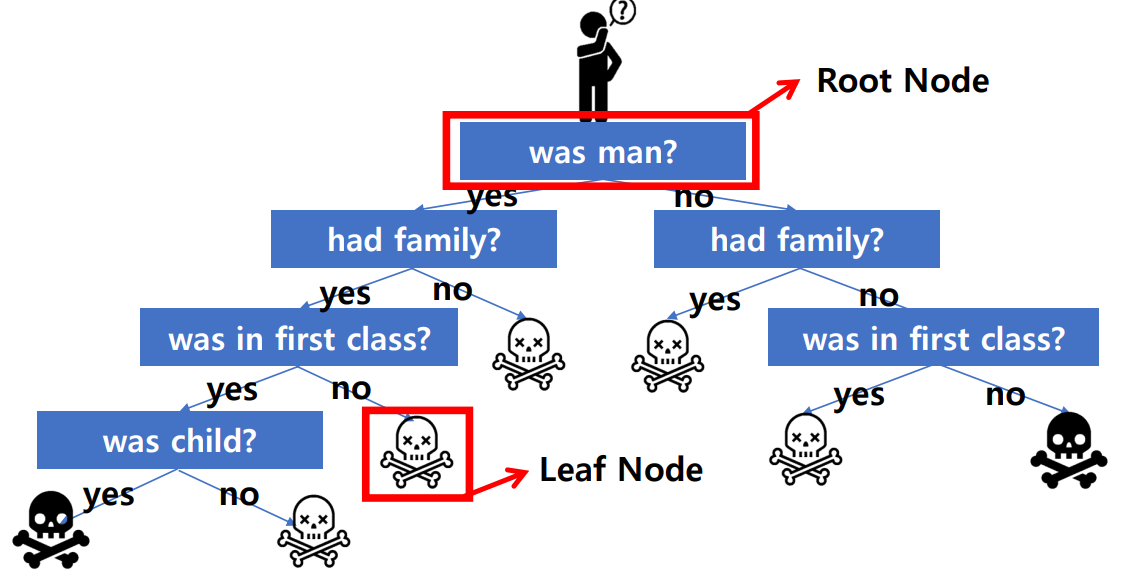
- 타깃 값이 한 개인 리프 노드를 순수 노드라고 한다.
- 모든 노드가 순수 노드가 될 때 까지 학습하면 복잡해지고 과대적합이 된다.
결정 트리 과대적합 제어
- 노드 생성을 미리 중단하는 사전 가지치기(pre-pruning)와 트리를 만든후에 크기가 작은 노드를 삭제하는 사후 가지치기(pruning)가 있다 (sklearn은 사전 가지치기만 지원)
- 트리의 최대 깊이(max_depth, 값이 클수록 모델의 복잡도가 올라간다)나 리프 노드의 최대 개수(max_leaf_nodes)를 제어
- 노드가 분할하기 위한 데이터 포인트의 최소 개수(min_samples_leaf)를 지정
지니 불순도
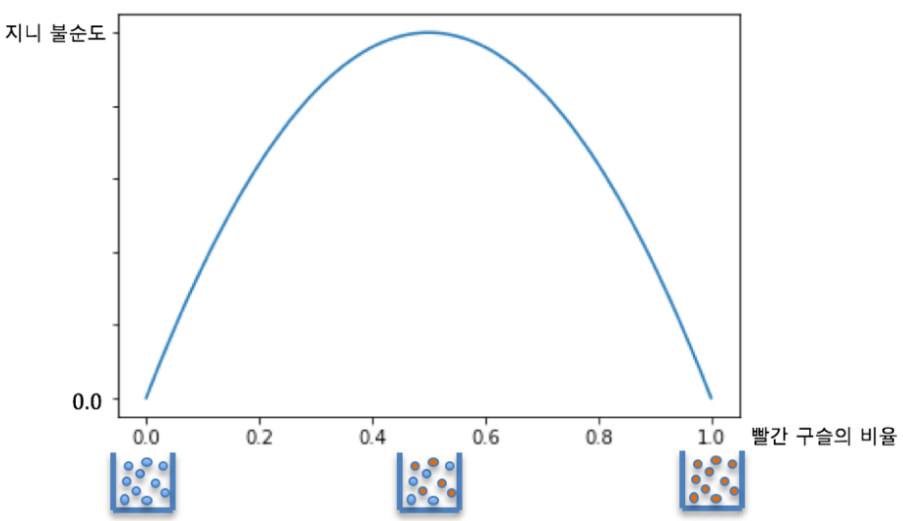
- 지니지수는 얼마나 불확실한가? (=얼마나 많은 것들이 섞여있는가?)를 보여준다.
- 지니 지수가 0이라는 것은 불확실성이 0이라는 것으로 같은 특성을 가진 객체들끼리 잘 모여있다는 의미이다.
- 지니 지수가 0.5(최대)라는 것은 반반 섞여 있다는 의미
Decision Tree 실습
목표
- 독버섯과 식용버섯을 분리하는 tree 모델 만들기
- tree 모델 시각화
- tree 모델의 특성 중요도 확인
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.metrics import accuracy_score
from sklearn.model_selection import train_test_split
- 데이터 불러오기
data = pd.read_csv('./data/mushroom/mushroom.csv')
display(data.shape)
display(data.head())
(8124, 23)
| poisonous | cap-shape | cap-surface | cap-color | bruises | odor | gill-attachment | gill-spacing | gill-size | gill-color | … | stalk-surface-below-ring | stalk-color-above-ring | stalk-color-below-ring | veil-type | veil-color | ring-number | ring-type | spore-print-color | population | habitat | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | p | x | s | n | t | p | f | c | n | k | … | s | w | w | p | w | o | p | k | s | u |
| 1 | e | x | s | y | t | a | f | c | b | k | … | s | w | w | p | w | o | p | n | n | g |
| 2 | e | b | s | w | t | l | f | c | b | n | … | s | w | w | p | w | o | p | n | n | m |
| 3 | p | x | y | w | t | p | f | c | n | n | … | s | w | w | p | w | o | p | k | s | u |
| 4 | e | x | s | g | f | n | f | w | b | k | … | s | w | w | p | w | o | e | n | a | g |
5 rows × 23 columns
data.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 8124 entries, 0 to 8123
Data columns (total 23 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 poisonous 8124 non-null object
1 cap-shape 8124 non-null object
2 cap-surface 8124 non-null object
3 cap-color 8124 non-null object
4 bruises 8124 non-null object
5 odor 8124 non-null object
6 gill-attachment 8124 non-null object
7 gill-spacing 8124 non-null object
8 gill-size 8124 non-null object
9 gill-color 8124 non-null object
10 stalk-shape 8124 non-null object
11 stalk-root 8124 non-null object
12 stalk-surface-above-ring 8124 non-null object
13 stalk-surface-below-ring 8124 non-null object
14 stalk-color-above-ring 8124 non-null object
15 stalk-color-below-ring 8124 non-null object
16 veil-type 8124 non-null object
17 veil-color 8124 non-null object
18 ring-number 8124 non-null object
19 ring-type 8124 non-null object
20 spore-print-color 8124 non-null object
21 population 8124 non-null object
22 habitat 8124 non-null object
dtypes: object(23)
memory usage: 1.4+ MB
X = data.iloc[:, 1:]
y = data['poisonous']
X.shape, y.shape
((8124, 22), (8124,))
- 데이터 전체를 원핫인코딩
X_onehot = pd.get_dummies(X)X_onehot.head()
| cap-shape_b | cap-shape_c | cap-shape_f | cap-shape_k | cap-shape_s | cap-shape_x | cap-surface_f | cap-surface_g | cap-surface_s | cap-surface_y | … | population_s | population_v | population_y | habitat_d | habitat_g | habitat_l | habitat_m | habitat_p | habitat_u | habitat_w | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | … | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | … | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | … | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| 3 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | … | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| 4 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | … | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
5 rows × 117 columns
X_train, X_test, y_train, y_test = train_test_split(X_onehot, y, test_size=0.3, random_state=926)
X_train.shape, X_test.shape, y_train.shape, y_test.shape
((5686, 117), (2438, 117), (5686,), (2438,))
- 모델 생성 및 학습
from sklearn.tree import DecisionTreeClassifier
tree_model = DecisionTreeClassifier()
tree_model.fit(X_train, y_train)
pred = tree_model.predict(X_test)
- 모델 평가
accuracy_score(pred, y_test)
1.0
- 트리의 특성 중요도 확인
- 트리 모델은 각 특성의 중요도를 확인 할 수 있음
- tree_model.feature_importances_
# 데이터프레임화 하기, 중요도 기준으로 내림차순 정렬
df = pd.DataFrame([X_train.columns, tree_model.feature_importances_]).T
df.sort_values(by=1, ascending=False)
| 0 | 1 | |
|---|---|---|
| 27 | odor_n | 0.615161 |
| 53 | stalk-root_c | 0.16913 |
| 55 | stalk-root_r | 0.093209 |
| 100 | spore-print-color_r | 0.034151 |
| 33 | gill-spacing_c | 0.024172 |
| … | … | … |
| 39 | gill-color_g | 0.0 |
| 38 | gill-color_e | 0.0 |
| 37 | gill-color_b | 0.0 |
| 34 | gill-spacing_w | 0.0 |
| 116 | habitat_w | 0.0 |
117 rows × 2 columns
Graphviz 시각화
- tree 모델 시각화
- Gini Impurity(지니 불순도) 확인해보기
from sklearn.tree import export_graphvizimport graphviz
export_graphviz(tree_model, # 저장할 트리 모델 객체
out_file='tree.dot', # 결과로 저장할 파일
class_names=['독', '식용'], # 클래스 이름 설정
feature_names=X_train.columns, # 컬럼 이름 넣어주기
impurity=True, # 불순도 표기 여부
filled=True, # 색상 채우기 여부
rounded=True # 수치값 반올림 여부
)
# 저장한 시각화 파일 불러오기
with open('./tree.dot', encoding='utf-8') as f:
dot_graph = f.read()
display(graphviz.Source(dot_graph))


댓글남기기